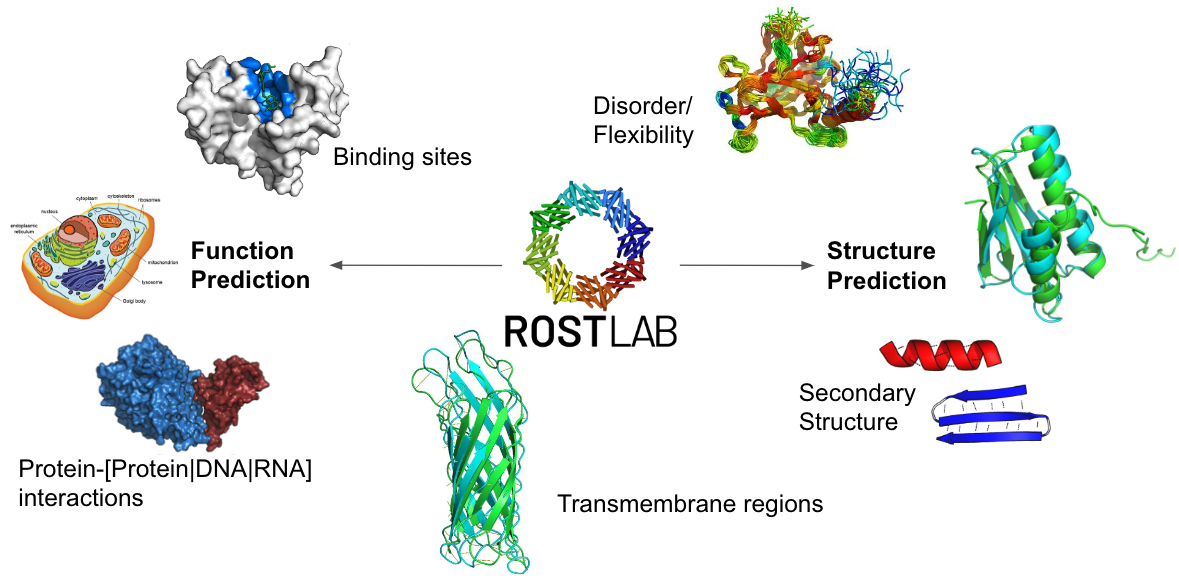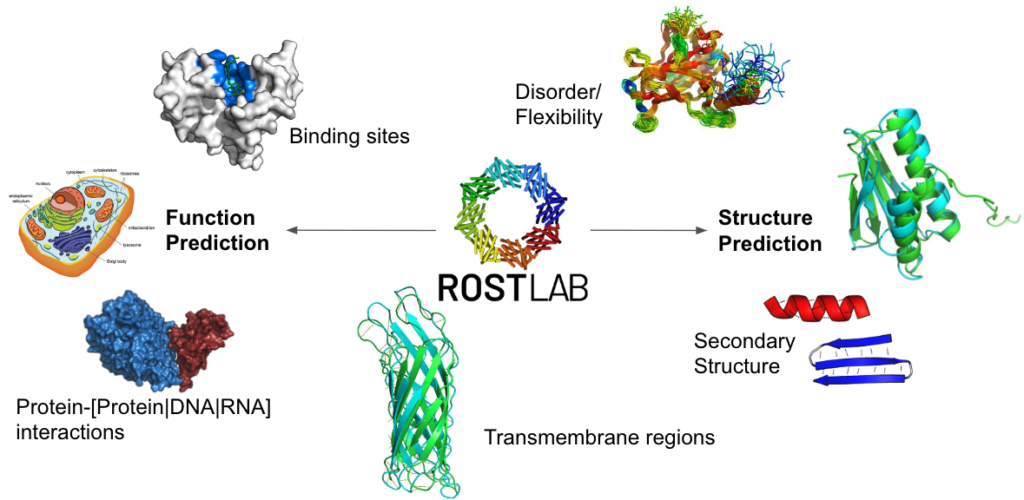
Das Ziel unserer Gruppe ist es, Aspekte der Proteinstruktur und -funktion anhand ihrer Sequenz vorherzusagen. Der enorme Reichtum an evolutionären Informationen, der durch den Vergleich der gesamten Biodiversität der Spezies zugänglich wird, macht dieses ehrgeizige Ziel erreichbar. Unsere besondere Nische liegt in der Kombination von evolutionären Informationen (EI) mit maschinellem Lernen (ML) und künstlicher Intelligenz (KI). Vor 30 Jahren ermöglichte die Verbindung von maschinellem Lernen und evolutionären Informationen (in Form von Mehrfachsequenzalignments) einen Durchbruch in der Vorhersage der Sekundärstruktur. Dasselbe Prinzip liegt allen modernen Vorhersagen von Proteinstruktur und -funktion zugrunde und ist auch der Ursprung des Programms, das einen Durchbruch in der Vorhersage von Proteinstrukturen erzielte, nämlich AlphaFold2. In den letzten zwei Jahren ist es möglich geworden, durch Protein-Sprachmodelle (pLMs) die Sprache des Lebens, die in Proteinen geschrieben ist, tiefgehend zu erlernen. Die extrahierten Informationen werden durch Transfer-Learning auf überwachte Lernmethoden für Proteinvorhersagen mit Anmerkungen übertragen.
Ich werde drei neue Methoden vorstellen, die die Proteinstruktur (1D: Sekundärstruktur, Membranregionen und Unordnung, 2D: Abstände/Kontakte zwischen Resten, 3D: Koordinaten) und die Proteinfunktion (subzelluläre Lokalisation, Bindungsstellen, GO-Terme) sowie die Auswirkungen von Sequenzvariationen unter Verwendung von pLMs vorhersagen. Diese Einbettungen ermöglichen es einigen Anwendungen, den aktuellen Stand der Technik zu erreichen oder sogar zu übertreffen, ohne evolutionäre Informationen zu nutzen. Entscheidend dabei ist das Verständnis der KI und die Kontrolle von Datenbankverzerrungen. In beiden Bereichen könnte die computergestützte Biologie als Experimentierfeld dienen, um empfindlichere KI-Anwendungen in der Gesellschaft vorzubereiten.
Mitmachstation:
Papierfaltmodelle z.B.: DNA Stränge, Viren, Proteine
Vortrag
Von Toxinen zu Therapeutika: Wie die Bioinformatik Schlangengifte für die menschliche Gesundheit nutzbar macht.
In diesem Vortrag werden die faszinierenden Möglichkeiten erforscht, wie giftige Substanzen, insbesondere Schlangengifte, durch den Einsatz der Bioinformatik zu wertvollen Werkzeugen in der Medizin und Forschung werden können. Giftstoffe, die von Schlangen, Spinnen oder anderen Tieren produziert werden, enthalten eine Vielzahl von bioaktiven Molekülen, die potenziell tödlich wirken können, aber auch ein enormes therapeutisches Potenzial bergen.
Vortrag ist auf englisch!
Standort
Boltzmannstr. 3
85748 Garching
TUM Mathematik & Informatik
Seminarraum 01.09.034